07 Avr La conception informatisée de protéines : Un chemin vers un traitement contre le coronavirus
Une demande de soutien financier pour les recherches du Pr Sarel Fleishman
Résumé
La maladie infectieuse COVID-19 associée au coronavirus SARS-CoV-2 représente la pandémie globale la plus dangereuse depuis la grippe espagnole du début du 20ème siècle. Afin de freiner la progression de ce coronavirus, les scientifiques ont entamé une course contre la montre pour trouver un traitement, de nouveaux outils de diagnostics et un vaccin pouvant prévenir une future propagation de cette infection.
Une approche générale développée par le professeur Sarel Fleishman, du département de science biomoléculaire de l’Institut Weizmann des Sciences, a récemment démontré la faisabilité d’un protocole qui pourrait accélérer le développement d’un vaccin contre le SARS-CoV-2. Cette approche, fondée sur plusieurs outils de conception de protéines par ordinateur développés dans le laboratoire du Pr Fleishman, pourrait représenter un moyen d’arrêter la pandémie actuelle et sauver des vies.
En se basant sur des algorithmes originaux fondés sur notre compréhension du comportement des protéines – comment elles se replient, se lient et interagissent – et qui prédisent la façon dont les mutations affectent leurs propriétés au niveau atomique, l’approche du professeur Fleishman permet de concevoir des anticorps fonctionnels hautement spécifiques – les « composants actifs » des traitements d’immunothérapie utilisés pour combattre les infections. Cette approche informatique offre, pour l’ingénierie d’anticorps, une alternative rapide et rentable aux méthodes utilisées dans la plupart des laboratoires de science de la vie, qui ont souvent des résultats non optimums.
Afin de faire progresser efficacement ce programme d’une importance cruciale, l’équipe du professeur Fleishman a besoin d’exploiter une quantité considérable de ressources de calcul, et ce très rapidement. Plutôt que d’améliorer les infrastructures informatiques à haut rendement présentes sur le campus – ce qui impliquerait une acquisition de matériel étalée sur plusieurs mois – les scientifiques ont prévu de mener leurs expériences directement sur le cloud d’Amazon. Cela permettra d’utiliser la puissance informatique de millions d’heures de calcul disponibles immédiatement et permettra de générer une base de données initiale de centaines de millions de modèles d’anticorps candidats. À partir de cette base de données il est prévu de mettre en place une plateforme expérimentale comprenant les différents stades de la biosynthèse d’anticorps, ainsi qu’un criblage à haut débit (ou sélection ultra-rapide) des candidats les plus efficaces. Ces candidats seront testés par les collaborateurs pour vérifier leur capacité à bloquer l’infection au SARS-CoV-2 fournissant ainsi les bases d’un futur traitement contre le COVID-19.
Afin de soutenir le professeur Fleishman et son équipe dans leur quête de la conception d’anticorps candidats comme base d’un traitement contre le coronavirus, l’Institut Weizmann des Sciences sollicite un don philanthropique d’un million de dollars.
Présentation du projet
Biologiste structural et novateur dans le domaine des technologies, le professeur Fleishman a développé un outil de modélisation informatique qui permet la conception rapide et peu coûteuse de protéines personnalisées aux propriétés attractives. La plus grande partie de son travail de conception se concentre sur les anticorps – les protéines qui, en s’attachant fermement à eux, nous défendent contre les virus et les bactéries pathogènes. Au cours de la crise actuelle, son laboratoire s’est concentré sur un élément structural particulier du coronavirus : les « protéines d’attachement » qui jouent un rôle clef dans le processus d’infection en permettant au virus de s’attacher aux cellules qu’il va infecter. Ces protéines ont été récemment décrites par des scientifiques de l’Université du Texas à Austin. Le but du professeur Fleishman est de concevoir un anticorps qui se lierait en des points vulnérables des protéines d’attachement et stopperait ainsi l’infection dans sa lancée.
Concevoir des anticorps capables de neutraliser un virus invasif est un défi informatique complexe
Une approche qui s’est avérée efficace contre le paludisme
L’impact potentiel de cette approche conceptuelle a été récemment démontré par une percée accomplie par le laboratoire du professeur Fleishman contre un autre tueur infectieux : le parasite Plasmodium falciparum qui cause le paludisme.

Le professeur Sarel Fleishman conçoit des protéines fonctionnelles qui combattent les maladies.
Il y a deux ans, un algorithme créé par le docteur Adi Goldenzweig – alors en doctorat dans le laboratoire du professeur Fleishman – a permis la conception d’un candidat intéressant pour un vaccin anti-paludisme. Non seulement les protéines synthétiques obtenues provoquent une réponse immunitaire de protection qui court-circuite l’infection par le parasite du paludisme, mais leur production est bon marché et elles restent stables à très haute température – un avantage significatif pour un vaccin conçu principalement pour les populations défavorisées vivant sous un climat tropical. Testées avec succès dans des études de laboratoire par les collègues du professeur Fleishman, ces nouvelles protéines paraissent prometteuses sur le terrain et sont actuellement en cours de dimensionnement pour créer un vaccin commercialisable.

Le docteur Adi Goldenzweig
Le défi : la complexité du coronavirus
Cependant, les anticorps qui permettront de bloquer parfaitement le SARS-CoV-2 ne seront pas faciles à concevoir. En effet, des travaux récents effectués par le professeur Jason McLellan de l’Université du Texas à Austin – un biologiste structural qui collabore avec l’équipe du professeur Fleishman de l’Institut Weizmann – ont apporté une information cruciale sur la structure du SARS-CoV-2. Une information qui va aider à définir des « cibles » informatiques pour la conception d’anticorps.
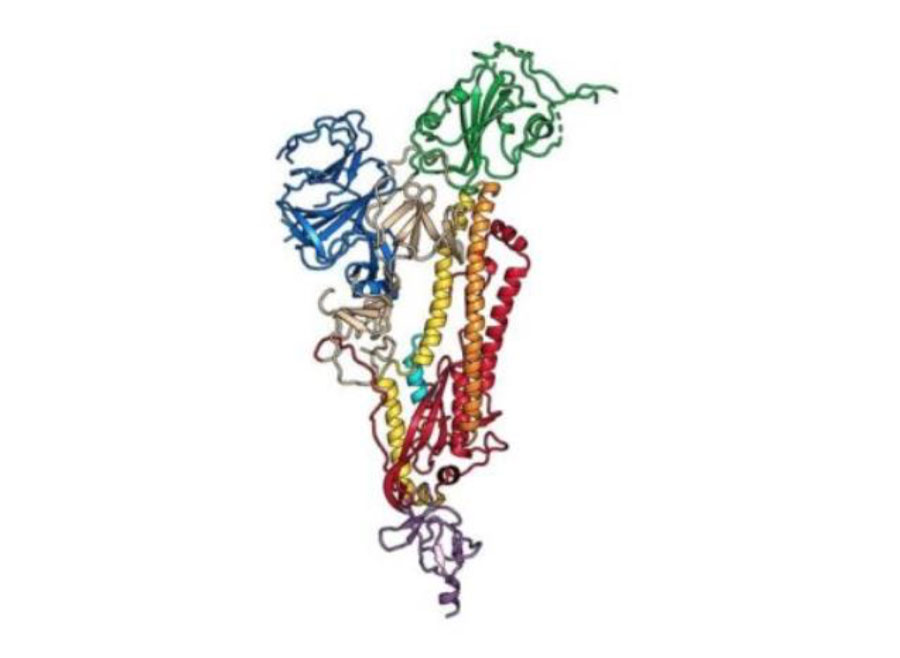
Cette image représente la structure de la « protéine d’attachement » qu’utilise le virus pour s’introduire de force dans les cellules
Le professeur McLellan et son équipe ont créé la première carte atomique 3D des protéines d’attachement du virus. Cette cartographie a révélé, sur ces protéines, des sites de vulnérabilité où des liants – comme les anticorps qu’espère concevoir le professeur Fleishman – pourraient potentiellement se fixer et neutraliser le mécanisme d’infection du virus. Mais malheureusement, ces sites vulnérables sont protégés par d’autres structures particulièrement complexes appelées glycanes.
Membres de la famille des polysaccharides, les glycanes sont de grandes molécules dans lesquelles un nombre important de molécules de sucre sont liées les unes aux autres. Leur structure complexe leur permet de jouer un rôle dans de multiples dynamiques moléculaires, notamment le développement et la différenciation de cellules immunitaires T, la reconnaissance de pathogènes et la signalisation immunitaire. Comme les glycanes recouvrent en partie les sites vulnérables des protéines d’attachement du coronavirus, les anticorps conçus pour atteindre et se lier à ces sites doivent être capables de se faufiler entre les glycanes pour atteindre leur cible. Or, si la nature nous fournit de nombreux exemples de protéines capables de se lier sélectivement et fortement à leur cible, ces protéines sont en général incroyablement complexes. Par exemple, une enzyme classique, ou la région d’un anticorps qui se lie à un antigène, est constituée de plus de 200 acides aminés et se replie en une conformation tridimensionnelle complexe qui dépend de milliers d’interactions atomiques. Ces molécules sont bien trop encombrantes pour passer entre les glycanes et atteindre les sites vulnérables des protéines d’attachement.
Cela rend le défi de la conception d’un anticorps encore plus complexe.
Une réponse au défi : le projet « nanocorps »
C’est pour contourner l’obstacle des glycanes que le professeur Fleishman a développé la première plateforme au monde capable de concevoir des « nanocorps » – de petites structures anticorps prêtes à se lier à de petits sites cibles avec une très grande précision.
Il prévoit d’étendre massivement un processus informatique qu’il a déjà validé à plus petite échelle dans son laboratoire. En utilisant les infrastructures informatiques très performantes de l’Institut Weizman et les algorithmes de conception de protéines développés dans son équipe (constituée de Lucas Krauss étudiant en doctorat, du docteur Ravit Netzer et du docteur Goldenzweig), le professeur Fleishman prévoit de concevoir des millions de nanocorps, tous optimisés pour se lier aux sites vulnérables du coronavirus. Le laboratoire synthétisera ensuite ces millions de nanocorps conçus informatiquement – ce qui, en soi, est un exploit rendu possible par les méthodes récemment développées dans le laboratoire – puis sélectionnera empiriquement, au sein de cet ensemble massif de données, les candidats les plus prometteurs en utilisant une technologie de tri à haut débit. Ce processus, qui devrait prendre quatre à six semaines, lui permettra d’identifier les anticorps présentant la plus grande affinité avec les sites cibles, c’est-à-dire les petites protéines présentant la combinaison la plus fiable de caractéristiques qui favoriseront une liaison étroite et spécifique avec les sites cibles – comme le choix de la bonne clef pour ouvrir un cadenas.
Les « finalistes » seront ensuite testés pour leur capacité à neutraliser les échantillons de coronavirus infectieux – une démonstration qui pourrait constituer la base d’un futur traitement du COVID-19 et aider au développement d’un vaccin.
Les atouts de l’Institut Weizmann : une plateforme, un pipeline et un plan
La stratégie mise en œuvre par le professeur Fleishman est parfaitement adaptée à la conception de nanocorps capables d’atteindre et de se lier aux sites vulnérables du virus qui, s’ils sont correctement ciblés, pourraient empêcher l’infection par le coronavirus.
Une expérience qui a fait ses preuves
L’équipe du professeur Fleishman a développé les premières méthodes entièrement automatisées permettant d’optimiser les fonctions de différentes protéines. En utilisant ces méthodes, ils ont conçu et produit une protéine qui est une candidate très intéressante pour un vaccin contre le paludisme.
Plus récemment son laboratoire a démontré comment améliorer considérablement des protéines thérapeutiques, en concevant une enzyme qui pourrait décomposer les agents nerveux toxiques – comme le sarin, le soman ou le VX russe – jusqu’à 4.000 fois plus efficacement qu’une enzyme classique, atteignant ainsi les niveaux nécessaires pour obtenir une protection thérapeutique.
Les méthodes innovantes du professeur Fleishman ont été relayées sur des serveurs web publics et ont été adoptées par des centaines de laboratoires dans le monde. Ces serveurs web, chacun focalisé sur un défi différent lié à la conception de protéines, sont disponibles gratuitement pour les groupes de recherches. Ces outils automatisés ont permis des découvertes dans divers domaines de la science fondamentale et clinique, faisant du professeur Fleishman l’un des chercheurs les plus influents du monde dans son domaine.
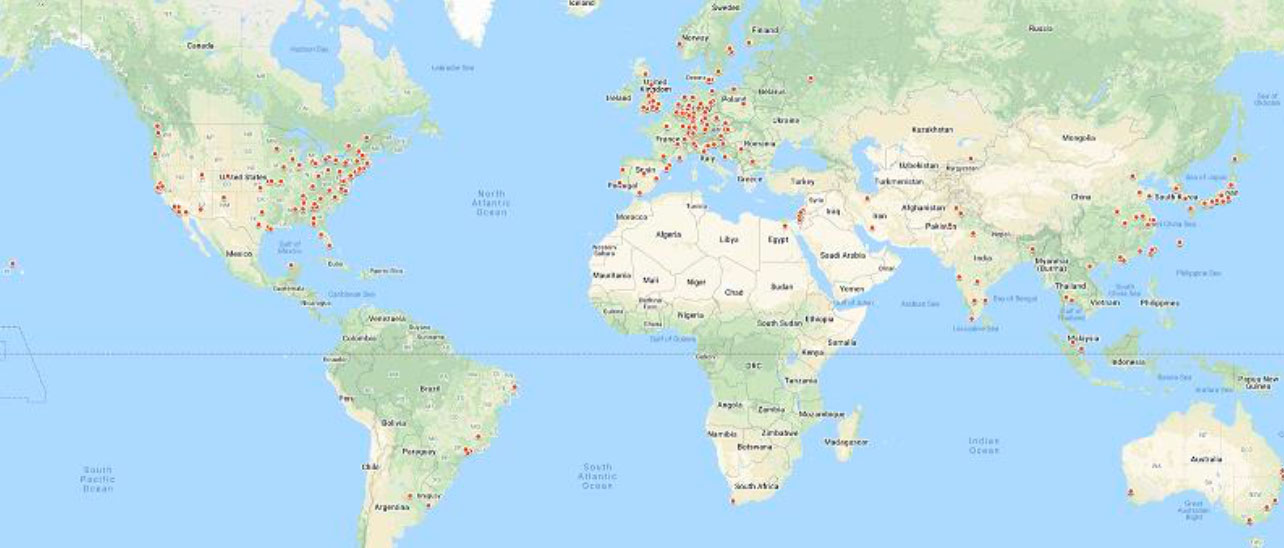
Les outils de conception de protéines du professeur Fleishman sont utilisés par des scientifiques partout dans le monde, comme l’indiquent les points rouges sur cette carte.
Des outils originaux de pointe :
Le professeur Fleishman et son équipe ont développé les outils algorithmiques ci-dessous. Ils sont disponibles en ligne gratuitement et participent aux avancées scientifiques du monde entier.
PROSS – Protein Repair One-Stop Shop(en français : guichet unique de réparation des protéines)
PROSS est un algorithme qui assiste les chercheurs qui travaillent avec des protéines complexes. Basé sur des structures et des séquences de protéines, PROSS génère de nombreuses séquences mutées afin de rendre les protéines plus stables.
TopGraph
TopGraph modélise la topologie des membranes en se basant sur l’orientation des segments qui les recouvrent. C’est un facteur déterminant pour réussir la conception d’une protéine.
AbPredict
AbPredict permet aux chercheurs de produire des modèles de structures d’anticorps de haute précision pouvant être utilisés pour les calculs de logiciels de conception comme Rosetta (modélisation de macromolécules).
FuncLib
Outil d’automatisation pour la création de bibliothèques d’enzymes, FuncLib permet de produire des jeux d’enzymes diverses de façon très précise, sans passer par les procédures de validation par un criblage à haut débit.
AffiLib
Cet outil identifie les stratégies permettant de concevoir des protéines avec l’affinité la plus proche de celle recherchée (par exemple, une protéine qui se lie plus étroitement et spécifiquement à une cible moléculaire).
AbLift
Cet automate utilise les données des mutations qui accentuent les affinités. Il permet d’accélérer la conception d’anticorps stables, avec une forte affinité avec les sites cibles. Il est utilisé dans la recherche et les applications cliniques.
Un leadership : le professeur Sarel Fleishman du département de science biomoléculaire
Le professeur Sarel Fleishman est né en Israël. Il a suivi le Programme interdisciplinaire d’excellence Adi Lautman et a effectué un travail de recherche en sciences de la vie à l’Université de Tel Aviv en 2000. Il a ensuite obtenu un master en sciences summa cum laude en 2002 puis un doctorat avec distinction en 2006, tous les deux à l’Université de Tel Aviv. Il a travaillé comme postdoctorant à l’Université de Washington (Seattle) de 2007 à 2011 puis a rejoint l’Institut Weizmann. Jusqu’à sa nomination au titre de professeur, le professeur Fleishman détenait la chaire Martha S. Sagon pour le développement de carrière. Il est le directeur de l’Institut du docteur Barry Sherman pour la chimie médicale.
Le professeur Fleishman a été plusieurs fois récompensé. En 2020, il a reçu le prix Hestrin de la Société israélienne de biochimie, en 2018 le prix du conseil scientifique de l’Institut Weizmann, en 2017 le prix Henri Guttwirth pour la recherche, en 2012 une bourse Alon du Conseil israélien pour l’enseignement supérieur, de 2006 à 2009 une bourse du Programme scientifique Human Frontier pour son postdoctorat, une bourse du Rothschild Postdoctoral Fellowship jusqu’en 2006, en 2008 le prix de recherche en biologie moléculaire et de 2003 à 2006 la bourse Sir Charles Clore pour son doctorat.
Besoins financiers
Afin de permettre au professeur Fleishman et à son équipe de continuer la conception d’anticorps candidats utilisés comme base d’un traitement contre le coronavirus, l’Institut Weizmann des Sciences sollicite un don philanthropique d’un million de dollars. Différentes modalités de reconnaissance seront proposées.
A propos de l’Institut Weizmann des Sciences
L’Institut Weizmann des Sciences à Rehovot, Israël, est l’un des meilleurs instituts au monde de recherche pluridisciplinaire. Reconnu pour son exploration diversifiée des sciences naturelles et exactes, l’Institut rassemble plus de 3 800 scientifiques, étudiants, techniciens et personnels de support. Les recherches effectuées à l’Institut s’orientent notamment vers de nouvelles façons de combattre les maladies ou la faim dans le monde, l’examen de questions mathématiques et informatiques, l’exploration de la physique de la matière et de l’univers, la création de matériaux innovants et le développement de nouvelles stratégies pour protéger l’environnement.
Institut Weizmann des Sciences
Rehovot 76100, Israël
resource.development@weizmainn.ac.il
www.weizmann.ac.il


