
06 Mai Des protéines sans parents
À l’aide de trois outils de prédiction des protéines par l’IA, une étude sino-israélienne met en évidence de nouveaux éléments dans l’histoire du repliement des protéines « orphelines ».
Lorsque les Prof. Joel Sussman et Israel Silman ont été invités à encadrer en ligne des étudiants chinois pendant la pandémie de COVID-19, ils ne s’attendaient pas du tout à ce que cette expérience débouche sur une recherche très innovante sur l’évolution des protéines, qui pourrait modifier notre compréhension de la manière dont de nouvelles protéines voient le jour.
« Au début, j’étais sceptique : il s’agissait d’étudiants de premier cycle et la communication via un écran d’ordinateur ne semblait pas très prometteuse », se souvient le professeur Sussman. Mais avec le professeur Silman – un duo de professeurs de l’Institut Weizmann des Sciences qui ont à leur actif des centaines d’études conjointes sur la structure et la fonction des protéines – ils ont accepté d’organiser des séances de tutorat pour une équipe de quatre étudiants issus d’universités de premier plan en Chine. Le mentorat en ligne faisait partie du programme YutChun-Weizmann, dirigé par le professeur Binghai Yan de l’institut Weizmann.
Les professeurs Sussman et Silman ont demandé aux étudiants de s’adresser à eux par leur prénom, une pratique inconnue dans les universités chinoises, et les ont encouragés à développer leur esprit critique. Pourtant, ils ne s’attendaient à rien de plus qu’un résumé respectueux lorsqu’ils ont demandé aux étudiants d’examiner un de leurs anciens articles sur les variations de la séquence des protéines. Au lieu de cela, les étudiants sont revenus avec une critique approfondie, analysant l’étude d’un point de vue contemporain et suggérant que certaines de ses conclusions pourraient être révisées à l’aide de nouvelles méthodes.

(de gauche à droite) Prof. Joel Sussman, Jing Liu et Prof. Israel Silman
Jing Liu, l’une des quatre mentorées, explique qu’il s’agissait, pour elle et les autres étudiants, d’un changement radical par rapport à ce à quoi ils étaient habitués. « En Chine, un étudiant en master, par exemple, ne peut pas contester un doctorant ou un post-doctorant – il pourrait se mettre en colère ou en parler au chercheur principal », explique-t-elle. Elle s’empresse toutefois de noter que l’environnement était différent sur le campus de Guangdong du Technion – Israel Institute of Technology, où elle étudiait à l’époque. « J’avais un superviseur qui était prêt à m’écouter et à discuter, ce qui est difficile à trouver dans d’autres universités en Chine.
À la surprise des deux parties, les travaux dirigés en ligne se sont rapidement transformés en discussions. Une étude réalisée en 2017 par des scientifiques tchèques, que Liu a portée à l’attention de ses tuteurs, est devenue l’un des principaux sujets de délibération – une étude qui laisse entrevoir un rebondissement intriguant dans l’histoire de l’évolution des protéines.
Des fissures dans le dogme du repliement
Au fur et à mesure que les organismes unicellulaires qui peuplaient autrefois la Terre évoluaient vers des organismes plus complexes, les modifications fortuites de leur ADN, si elles étaient bénéfiques, avaient tendance à être conservées, grâce à la sélection naturelle, et à être transmises aux organismes supérieurs. C’est pourquoi la plupart des gènes codant pour des protéines dans notre corps ont des équivalents (le terme scientifique est « homologues ») dans de nombreuses autres espèces le long de l’arbre de l’évolution, jusqu’à la levure ou la bactérie. Au fur et à mesure que les protéines se développaient, nombre d’entre elles ont commencé à se replier en structures complexes qui leur permettaient d’accomplir des tâches particulières.
Étant donné que la sélection naturelle est à l’œuvre depuis des milliards d’années, il semblerait que les protéines aient eu suffisamment de temps pour évoluer vers toutes les séquences utiles possibles. En fait, jusqu’à récemment, les scientifiques pensaient que toutes les protéines existantes étaient nées du raffinement de séquences existantes et que les protéines véritablement nouvelles avaient cessé d’apparaître depuis longtemps.
Mais il y a un peu plus d’une décennie, des fissures ont commencé à apparaître dans cet évangile scientifique : Il est apparu que de nouvelles protéines continuaient à naître en permanence. Lorsque les scientifiques ont commencé à séquencer des génomes entiers de divers organismes, les comparaisons ont révélé la présence de gènes codant pour des protéines « nouvellement nées » dans toutes les espèces, des bactéries à l’homme. On pense que ces protéines trouvent leur origine dans les régions non codantes qui constituent la majeure partie du génome. Dans ce scénario, une portion d’ADN dépourvue de message codant pour des protéines acquiert, par hasard, un ensemble de mutations qui la transforment en gène codant pour une protéine.
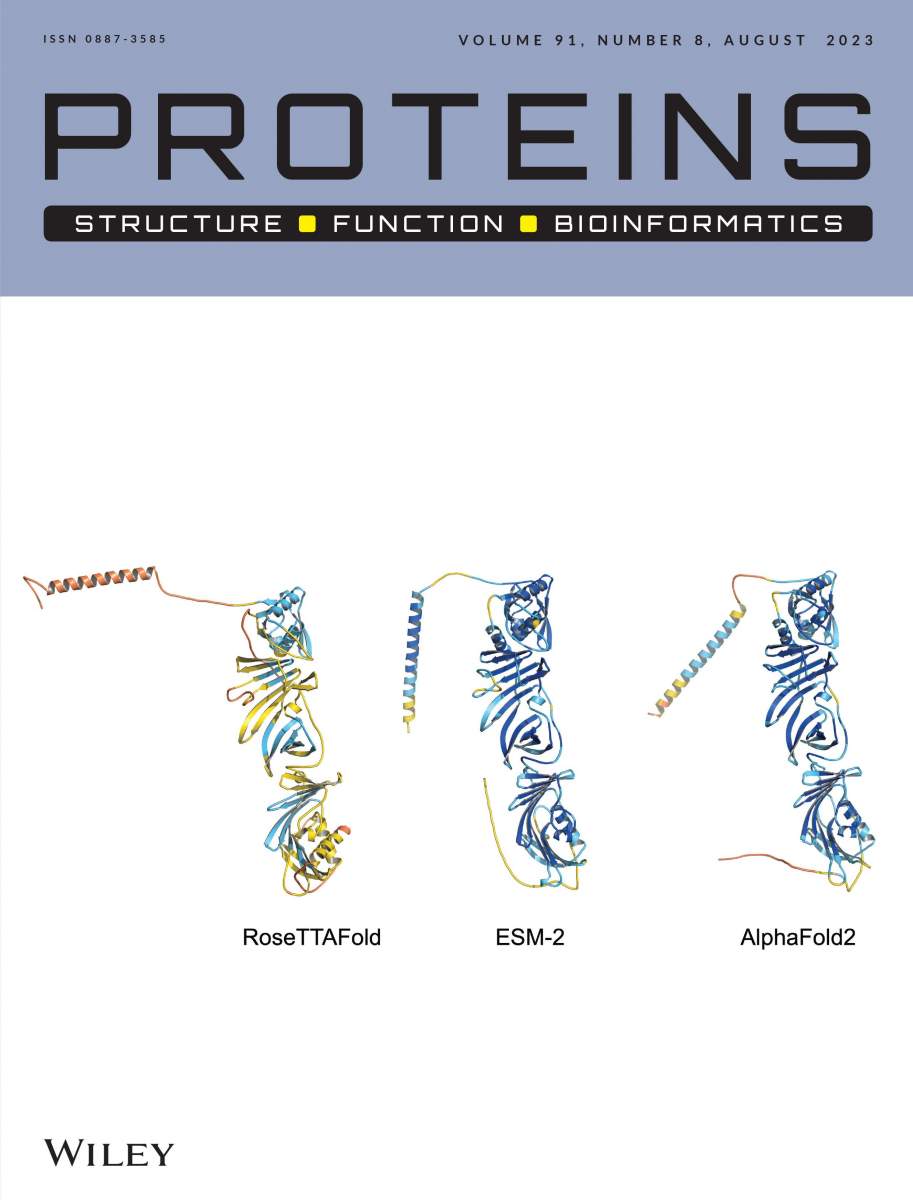
Les structures 3D d’une protéine « nouvellement née », prédites par trois outils d’intelligence artificielle différents, sur la couverture de la revue.
L’étude tchèque qui a tant intrigué Liu et ses tuteurs a ouvert une nouvelle brèche dans le dogme. Les chercheurs tchèques avaient créé une centaine de séquences de protéines hypothétiques en remaniant au hasard les gènes des protéines existantes, comme un jeu de cartes. Lorsqu’ils ont synthétisé ces protéines « jamais nées » et les ont testées en laboratoire, ils ont découvert qu’environ un tiers d’entre elles montraient des signes de repliement en structures compactes, un peu comme les protéines naturelles.
« C’était tout à fait étonnant », déclare le professeur Sussman. « Si quelqu’un m’avait demandé auparavant si une séquence de protéines aléatoires pouvait se replier de cette façon, j’aurais répondu que jamais.
Le professeur Silman explique que la capacité des protéines à se replier est essentielle à la vie. Bien que toutes les protéines ne se replient pas, ce sont celles qui sont repliées, c’est-à-dire celles dont les segments sont ordonnés, qui remplissent les fonctions catalytiques essentielles dans les organismes vivants. En montrant que des protéines « jamais nées » peuvent se replier, l’étude tchèque suggère que de nouvelles protéines peuvent non seulement naître, mais aussi jouer de nouveaux rôles vitaux.
Les orphelins de naissance
Comment un segment d’ADN non codant produit-il une protéine « nouvellement née » et comment cette protéine devient-elle active ? Quelle est l’échelle de temps de ces processus ? Et les mécanismes impliqués peuvent-ils être un jour exploités dans la conception de protéines ?
Pour répondre à ces questions, les professeurs Sussman et Silman ont décidé de mener ce qui, à leur connaissance, est devenu l’une des premières études structurelles de protéines nouvellement nées. Ils ont lancé le projet avec Jing Liu, premier auteur de l’article, et Rongqing Yuan, alors étudiant à l’université Tsinghua de Pékin. Les quatre se sont rencontrés en ligne pendant un an et demi avant de terminer la recherche, qui a été publiée récemment dans la revue Proteins : Structure, Function, and Bioinformatics. Les deux autres étudiants, Wei Shao et Jitong Wang, ont participé aux premières étapes du projet ; ils ont abandonné à la fin du tutorat prévu mais sont coauteurs de l’article publié.
L’équipe a exploré le potentiel de repliement des protéines « nouvellement nées » à l’aide d’outils d’intelligence artificielle (IA) qui, ces dernières années, ont révolutionné l’étude des structures protéiques. Dans la plupart des cas, ces algorithmes peuvent désormais prédire de manière fiable la structure tridimensionnelle d’une protéine sur la base de sa seule séquence d’acides aminés, sans qu’il soit nécessaire de cultiver des cristaux de protéines et de les utiliser pour déterminer par diffraction des rayons X la structure de la protéine cristallisée.

Rongqing Yuan
L’un des principaux défis auxquels l’équipe a été confrontée est que ces algorithmes de prédiction fonctionnent mieux lorsque la protéine en question a de nombreux homologues, c’est-à-dire des équivalents chez d’autres espèces, alors que les protéines « nouvellement nées », par définition, n’existent que chez une seule ou une poignée d’espèces. Comme elles n’ont pas de parents évolutifs, elles sont parfois appelées protéines orphelines (ou quasi-orphelines, si elles n’existent que dans quelques espèces apparentées). Il a fallu toute l’expertise de l’équipe pour appliquer avec succès les outils d’IA aux protéines orphelines sans homologues. Pour augmenter les chances d’obtenir des résultats fiables, les scientifiques ont utilisé trois algorithmes d’IA différents – AlphaFold2, RoseTTAFold et ESMFold – et ont comparé leurs résultats.
Tout d’abord, l’équipe a utilisé les trois algorithmes pour prédire les structures 3D des séquences de protéines « jamais nées » et mélangées de l’étude tchèque. Les prédictions ont permis d’identifier la structure de chaque protéine comme étant pliée ou désordonnée d’une manière qui correspondait aux résultats expérimentaux de l’étude.
Ensuite, Jing Liu, Rongqing Yuan et leurs mentors israéliens ont appliqué les algorithmes aux protéines orphelines « nouvellement nées », dont très peu avaient été purifiées et caractérisées expérimentalement de manière adéquate. Après avoir parcouru la littérature scientifique, les scientifiques ont identifié sept protéines orphelines dont la fonction, mais pas la structure, était connue.
Les outils d’IA ont indiqué que cinq des sept étaient pliés de manière compacte, tandis que deux semblaient ne pas avoir de structure définie. Pour l’une des cinq protéines, les trois algorithmes ont fait des prédictions tellement similaires – signalant une très grande probabilité d’exactitude – que la revue a présenté les trois structures 3D sur sa couverture.
En outre, les scientifiques ont consulté la banque de données des protéines et ont trouvé trois protéines orphelines dont la structure cristalline avait été déterminée expérimentalement. Fait remarquable, deux de ces protéines présentaient des plis dont l’existence n’est pas connue ailleurs. Étant donné que la structure détermine la fonction d’une protéine, les nouveaux plis suggèrent que certaines protéines orphelines pourraient remplir des fonctions biologiques inconnues jusqu’à présent qui, à l’avenir, pourraient être exploitées dans une foule d’applications utiles, allant de la dégradation des plastiques à la production d’énergie propre ou au traitement des maladies.
Selon le professeur Sussman, « cette recherche modifie notre idée de la manière dont l’évolution pourrait fonctionner. L’évolution progresse généralement de la manière décrite par Darwin, mais il arrive que des protéines apparaissent, en quelque sorte, de nulle part. Ainsi, de nouvelles caractéristiques pourraient surgir de nulle part, pour ainsi dire, plutôt que d’avoir évolué à partir d’ancêtres sur des millions d’années. « Le professeur Silman ajoute que les résultats de l’étude, ainsi que d’autres études sur les protéines « nouvellement nées », modifient la réflexion sur l’origine de la vie en général, et de l’homme en particulier : « Il semble que nous ne soyons pas seulement les arrière-petits-enfants d’E. coli.
Nous espérons que notre étude incitera d’autres scientifiques à examiner les protéines orphelines à l’aide d’outils de prédiction IA afin de se faire une idée de leur structure et de leur fonction », résume le professeur Sussman. Lorsqu’une structure entièrement nouvelle apparaît, tous les paris sont ouverts quant à la fonction biochimique de la protéine. C’est alors que s’ouvrent de nouveaux horizons de recherche passionnants ».
Jing Liu prépare actuellement sa maîtrise en sciences dans le laboratoire du professeur Naama Barkai au Département de Génétique Moléculaire de Weizmann, et Rongqing Yuan est actuellement étudiant diplômé à l’université du Texas Southwestern Medical Center, à Dallas. Le professeur Sussman fait partie du Département de Biologie Chimique et Structurelle de Weizmann, le professeur Silman du Département des Sciences du Cerveau et le professeur Binghai Yan du Département de Physique de la Matière Condensée. Le professeur Amit Finkler, du Département de Physique Chimique et Biologique, a coordonné le programme YutChun-Weizmann à la faculté de chimie.
Le programme YutChun-Weizmann fait partie d’une initiative visant à promouvoir la collaboration universitaire entre la Chine et la communauté scientifique internationale. Parmi ses activités, le programme offre à des étudiants de premier cycle exceptionnels des possibilités de recherche.
YutChun (雨春) est l’abréviation chinoise de la pluie (雨) au printemps (春), qui symbolise l’épanouissement de la nouvelle génération dans les domaines de la science et de la technologie.
La Science en Chiffres
On trouve au moins 12 protéines « nouvellement nées » biologiquement significatives chez les primates ; deux d’entre elles ne se trouvent que chez l’homme.


