
09 Mai La protéine porte du Prada
Une nouvelle méthode de calcul permettant de suivre les multiples modifications des protéines promet d’apporter un éclairage nouveau sur des problèmes biomédicaux non résolus.
De même qu’une nouvelle tenue vestimentaire peut nous faire assumer une autre personnalité, une étiquette chimique « portée » par une protéine peut rendre cette molécule méconnaissable. Toute une garde-robe d’étiquettes – appelées modifications de protéines, car elles modifient la structure chimique originale de la protéine – aide les milliers de protéines de notre corps à accomplir leurs tâches polyvalentes, mais ces modifications compliquent la vie des scientifiques et des médecins qui cherchent à identifier ces molécules vitales. Le laboratoire du professeur Yifat Merbl, à l’Institut Weizmann des Sciences, a relevé le défi de détecter simultanément un grand nombre de protéines modifiées, ainsi que tous les éléments qui les accompagnent.
Le port de ces étiquettes, connues sous le nom de modifications de protéines, peut changer la vie d’une personne comme si elle portait un costume de super-héros ou une tiare. Les étiquettes peuvent affecter presque toutes les propriétés essentielles des protéines, notamment leur stabilité, leur localisation dans la cellule et leur liaison à d’autres molécules. Elles peuvent également marquer les protéines malades pour qu’elles soient détruites ou déterminer la capacité d’une protéine, telle qu’un suppresseur de tumeur, à remplir sa fonction. C’est pourquoi la détection et l’analyse des protéines modifiées sont essentielles pour l’étude de divers processus corporels et maladies. Par exemple, l’identification des protéines qui ont été modifiées d’une manière propre aux cellules cancéreuses pourrait aider à repérer et à détruire ces cellules plus efficacement ; la recherche d’autres types de modifications anormales pourrait aider les scientifiques à expliquer et peut-être même à traiter les maladies auto-immunes, dans lesquelles le système immunitaire attaque par erreur les propres tissus de l’organisme.

De g. à d. : Assaf Kacen, Prof. Yifat Merbl et Merav D. Shmueli. Illuminer les modifications des protéines
Mais considérez l’ampleur du défi. Le génome humain compte quelque 20 000 gènes codant pour des protéines, dont chacun peut se décliner en quatre ou cinq versions, ce qui donne environ 100 000 séquences protéiques possibles. Une fois qu’une protéine est créée, elle peut être modifiée chimiquement par une ou plusieurs des quelque 200 étiquettes différentes. Si l’on ajoute à cela le fait que ces marqueurs peuvent se fixer à plusieurs endroits sur une molécule de protéine, on obtient un nombre pratiquement illimité de combinaisons de marquage possibles et, par conséquent, de protéines modifiées différemment.
Les méthodes standard ne peuvent pas reconnaître ces protéines modifiées. Chaque protéine possède une séquence unique de blocs d’acides aminés qui permet de l’identifier en comparant cette séquence à des plans de protéines dans des bibliothèques de référence, mais les modifications empêchent cette reconnaissance car elles altèrent les propriétés des acides aminés. L’analyse simultanée d’un grand nombre de ces modifications étant extrêmement difficile, une vaste étude des protéines modifiées – qui poserait des questions ouvertes – était trop ambitieuse. Ainsi, jusqu’à présent, la plupart des études ont eu tendance à se concentrer sur seulement quelques modifications spécifiques.
« Observer les effets des protéines sans tenir compte des modifications qui se produisent en elles après leur création limite notre compréhension de la véritable complexité des processus biologiques », explique le Prof. Merbl, du Département d’Immunologie des Systèmes de Weizmann. « Mon laboratoire s’est fixé pour objectif de créer un nouvel outil informatique capable de traquer des dizaines de types de modifications de protéines de manière non biaisée. »

À l’instar des minifigures Lego, les protéines peuvent revêtir diverses tenues – des étiquettes chimiques qui modifient leurs propriétés, ce qui les rend difficiles à reconnaître. La nouvelle méthode permet d’identifier une trentaine de modifications en six heures, alors que les méthodes existantes ne pouvaient en identifier que trois à la fois et prenaient environ deux semaines pour le faire
La stratégie développée par le Prof. Merbl et son équipe a été de rechercher de nombreuses altérations en parallèle, plutôt que de les rechercher individuellement, comme cela se faisait auparavant. Les scientifiques ont appelé leur système PROMISE (Protein Modification Integrated Search Engine), et il porte bien son nom : il peut identifier une trentaine de modifications en six heures, alors que les méthodes existantes ne pouvaient en identifier que trois à la fois et prenaient environ deux semaines pour y parvenir. Le développement du système a été dirigé par Assaf Kacen, en collaboration avec Aaron Javitt ; tous deux étaient doctorants dans le laboratoire du Prof. Merbl.
« PROMISE nous fournit de nouvelles lunettes, offrant aux chercheurs une image plus large des processus biologiques, et ce à une résolution plus élevée que jamais », déclare le Prof. Merbl. « Nous pouvons maintenant utiliser cet outil pour résoudre des problèmes biomédicaux non résolus en réexaminant différents types d’ensembles de données existants. »
Pour la première application de PROMISE, Kacen, Javitt et d’autres membres de l’équipe du Prof. Merbl ont décidé de s’attaquer à une question fondamentale concernant le cancer : si et comment les modifications des protéines affectent la capacité du système immunitaire à éliminer les tumeurs. Les chercheurs ont appliqué PROMISE pour examiner des centaines de milliers de peptides (fragments de protéines) provenant de plus de 200 échantillons de cellules et de tissus liés au cancer. En particulier, l’équipe s’est concentrée sur les peptides connus sous le nom d’antigènes tumoraux. Présents à la surface des cellules tumorales, les antigènes signalent au système immunitaire que ces cellules sont anormales et doivent être éliminées. Après avoir vérifié les échantillons pour une trentaine de combinaisons de modifications protéiques, les scientifiques ont réussi à identifier un nouveau sous-ensemble de peptides, environ 12 000 au total, contenant des modifications qui n’avaient pas été détectées auparavant par d’autres outils informatiques. Le résultat est l’atlas le plus complet disponible des antigènes modifiés caractéristiques des tumeurs cancéreuses. Il comprend un nouveau « dictionnaire », une référence montrant comment les modifications survenant à des sites spécifiques dans les peptides peuvent affecter la reconnaissance de la cellule cancéreuse par le système immunitaire.
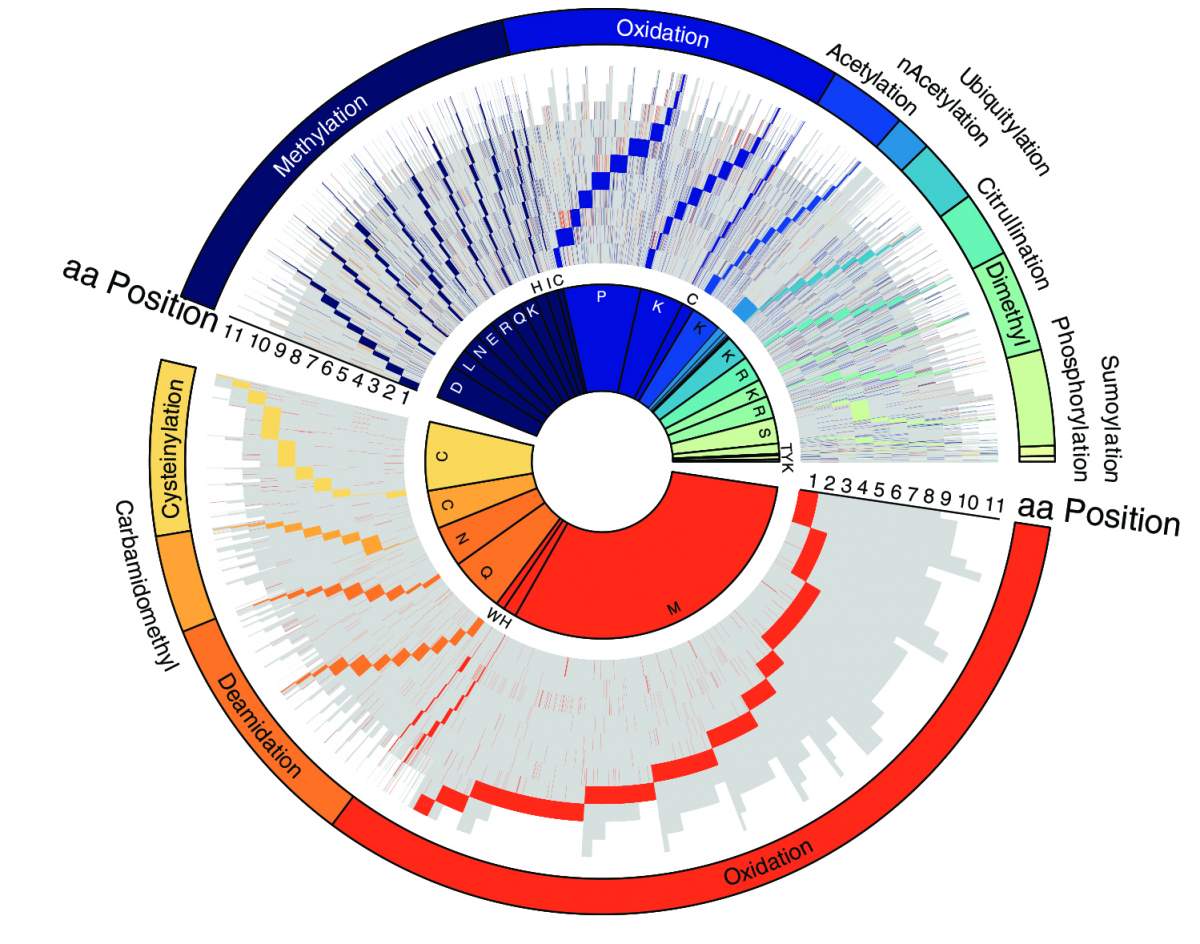
Représentation schématique des modifications protéiques dans quelque 12 000 peptides analysés par PROMISE, triés par type de modification et position dans le peptide. Chaque ligne représente un peptide distinct, en gris ; les sites des différentes modifications sont marqués par des couleurs.
Ces connaissances sont essentielles car certaines de ces modifications pourraient expliquer comment les cellules cancéreuses se « camouflent » et sont ensuite capables d’échapper aux lymphocytes T utilisés dans l’immunothérapie du cancer. Cette compréhension pourrait à son tour aider les chercheurs à mettre au point des méthodes de « décloisonnement » pour améliorer l’attaque ciblée contre la tumeur. Par exemple, une étiquette phosphorée, qui a une charge négative, peut permettre à un peptide cancéreux d’éviter d’être reconnu par les lymphocytes T, mais la conception de récepteurs de lymphocytes T capables de repérer de telles étiquettes pourrait accroître le potentiel thérapeutique des cellules.
Dans de futures études, PROMISE pourrait aider à révéler les combinaisons de modification peptidique qui incitent les cellules immunitaires à attaquer les cellules saines, provoquant ainsi des maladies auto-immunes. Cet outil pourrait également fournir de nouvelles informations sur les troubles neurodégénératifs et d’autres maladies.
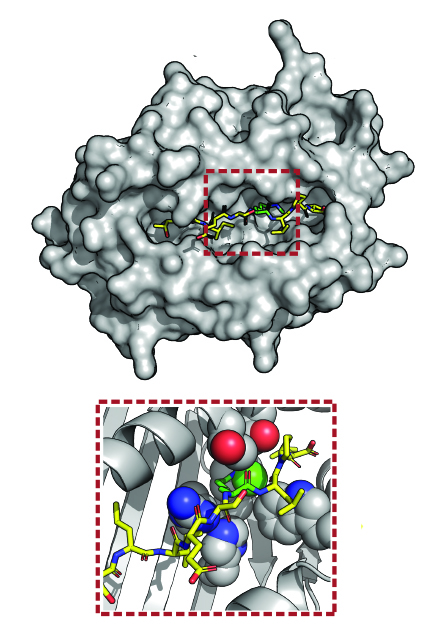
Modèle 3D d’un complexe protéique immuno-régulateur (en haut), avec un zoom avant (en bas) sur un peptide (jaune) contenant une modification (vert).
« Pouvoir analyser rapidement les modifications des protéines peut nous aider à mieux diagnostiquer de nombreux processus pathologiques, à évaluer la réponse aux médicaments et, en fin de compte, à développer de nouvelles thérapies », explique le Prof. Merbl.
La Science en chiffres
Plus de 10 % des protéines du génome humain sont des enzymes qui modifient chimiquement d’autres protéines. Les outils d’analyse standard, qui ne tiennent compte ni de ces modifications ni des mutations et autres écarts par rapport au schéma génétique, n’identifient que 50 à 80 % des peptides dans les échantillons biologiques. En d’autres termes, dans tout échantillon de tissu, jusqu’à 50 % des peptides peuvent ne pas être identifiés


